Analyse de données interopérables en oncologie : le modèle conceptuel OSIRIS
Mis à jour le
OSIRIS est l’acronyme de "GrOupe inter-SIRIC sur le paRtage et l’Intégration des donnéeS clinico-biologiques en cancérologie". Ce groupe de travail a établi un modèle conceptuel de données interopérable, du même nom, et est construit à partir d’outils repris de l’informatique décisionnelle.
À terme, l’objectif est d’implémenter OSIRIS dans un maximum de systèmes d’informations hospitaliers au niveau national afin de réaliser des projets de recherche utilisant les données de santé appliquées à l’oncologie.
Pourquoi utiliser le modèle OSIRIS ?
Santé et données
La science des données ("data science") appliquée à l’oncologie est un domaine récent qui fait appel à des compétences spécifiques en mathématiques, en informatique et en oncologie. Ses objectifs sont d’assurer :
- la description et l’analyse des données à disposition ;
- les diagnostics en oncologie ;
- l’analyse des causes possibles d’un cancer ;
- la prédiction d’évènements (récidives, …) ;
- l’aide à la prise de décision médicale.
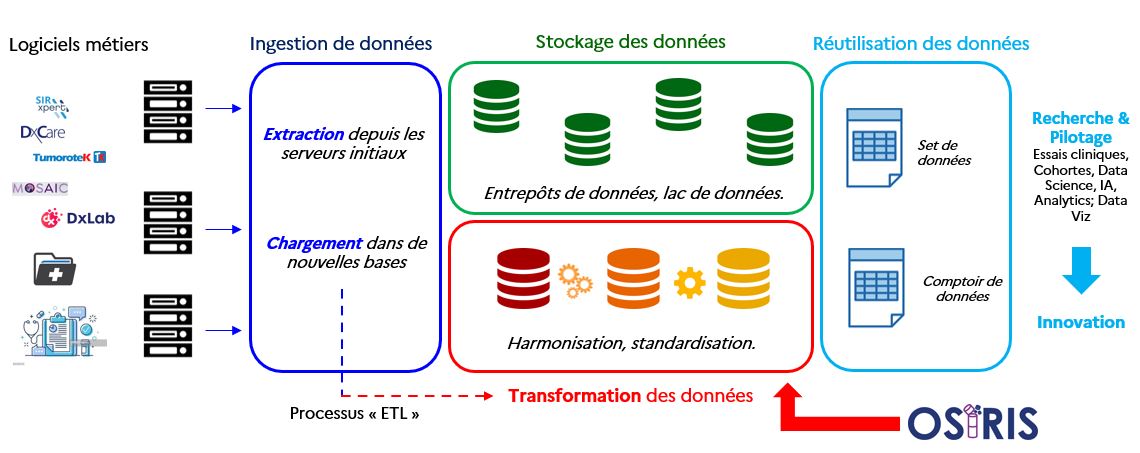
Dans un établissement de santé, des millions de données sont générées à partir de différents logiciels et équipements médico-administratifs. Pour certains de ces établissements, ces données sont ensuite regroupées dans des bases de données intermédiaires, puis extraites, transformées et chargées dans un entrepôt de données.
Le développement d'algorithmes en santé nécessite des données de qualité, normalisées et disponibles en grande quantité. Il est donc nécessaire de formater ces données pour répondre à différentes exigences de qualité. Le partage des données est également primordial pour générer des ensembles de données de grande taille afin d’alimenter des modèles algorithmiques.
De cette normalisation découlent les notions d'interopérabilité et de standardisation, qui désignent la capacité des systèmes d'information hospitaliers à communiquer entre eux selon un même langage.
Un besoin urgent de standardisation pour une mise à l’échelle efficace
Aujourd'hui, de nombreuses voix scientifiques s'élèvent aux niveaux national et international, appelant les pouvoirs publics à s'impliquer davantage dans l'élaboration de normes et de standards pour structurer les données de santé et les rendre interopérables (Source).
Par exemple, la majorité des projets d’intelligence artificielle échoue à l’étape d’industrialisation pour un certain nombre de raisons (Source : Pourquoi les projets de Machine Learning échouent) :
- Une sous-évaluation de l’effort requis pour collecter les données d’apprentissage nécessaires.
- Le manque de spécificité des données obtenues pour l’apprentissage (qualité et disponibilité des données).
- L’absence de flexibilité sur la capacité à modifier ses requêtes de données pour procéder à des ajustements concernant les données devant être collectées pour l’apprentissage.
- L’absence de vérification de la qualité et du sens des données d’apprentissage choisies.
- La mise en place de revue systématique des données et des algorithmes utilisés en production pour adapter leurs maintenances.
Le succès des futurs projets de recherche en données issues de l’oncologie reposera donc sur l’utilisation de standards permettant la collecte de données à grande échelle, de qualité et maintenus dans le temps.
Historique du projet OSIRIS
Le projet OSIRIS est une initiative menée par le groupe inter-SIRIC travaillant sur le partage de données cliniques ou génomiques pour répondre aux enjeux de la recherche sur le cancer en matière de science des données. Ces enjeux sont les suivants :
- la conceptualisation du cancer pour rendre exploitables les données de santé dans le cadre de questions de recherche en oncologie ;
- l’interopérabilité pour permettre l’échange de données entre établissements de santé selon des standards informatiques internationaux de communication ;
- la standardisation des données de santé pour générer des sets de données de qualité et éviter des erreurs d’exploitation et d’interprétation.
Dans le domaine de l'oncologie, le projet OSIRIS a été lancé en France il y a une dizaine d'années pour répondre à ces trois enjeux.
L'objectif d'OSIRIS est donc de modéliser le cancer à l'aide de variables choisies par les experts (conceptualisation), exprimées dans des terminologies et des unités validées par la communauté scientifique (standardisation) dans le cadre des données cliniques et des données génomiques, le tout dans un format de communication adéquat (interopérabilité).
Cette structuration des bases de données permet alors de partager des données standardisées entre différents établissements de santé, compréhensibles par tous les systèmes participants et facilitant l’exploitation des données dans le cadre d’essais cliniques ou de questions de recherche en oncologie. Les résultats de ce projet collaboratif OSIRIS, financé par l'Institut national du cancer à hauteur de 300 000€, ont été publiés dans le JCO. Clinical Cancer Informatics (Guerin et al, OSIRIS: a minimum dataset for data sharing and interoperability in oncology, 2021).
Ce travail a permis de sélectionner 130 variables cliniques et génomiques capables de représenter l'évolution de la maladie cancéreuse au fil du temps et de démontrer la faisabilité de l'interopérabilité technique par le biais d'une infrastructure de type fédérée interrogeant à distance les bases de données des centres participants.
MED-OSIRIS, projet dans la continuité d'OSIRIS
MED-OSIRIS (Maintenance, extension et déploiement du modèle OSIRIS) est un second projet dans la continuité du projet OSIRIS, également soutenu par l'Institut à hauteur de 150 000€ depuis 2021.
Le premier objectif est d'assurer la maintenance et la mise à jour du modèle de données OSIRIS initial au sujet des terminologies internationales de santé, des nouveaux éléments de données ou de nouveaux concepts de données. L'extension du projet consiste ensuite à travailler dans le domaine de l'imagerie médicale avec des experts additionnels dans le but d’étendre le modèle de données OSIRIS initial avec des concepts de données radiomiques et de radiothérapie. Enfin, le déploiement consiste à établir une correspondance entre le modèle de données commun OSIRIS et le standard FHIR, un standard de santé développé pour faciliter l'échange de données de santé électroniques entre différents systèmes d'information hospitaliers. Cette partie implique l'installation de la solution OSIRIS-FHIR dans des entrepôts locaux.
Le second objectif est de démontrer, à l'aide d'une étude de cas pilote, OSIRIS LUNG, la pertinence clinique du modèle conceptuel de données OSIRIS pour accélérer l'innovation. L'objectif principal est de mieux comprendre l'hétérogénéité de la réponse aux thérapies ciblées et d'identifier des facteurs prédictifs ou des biomarqueurs de la réponse au traitement en utilisant des données de vie réelle de patients atteints d'adénocarcinome pulmonaire dans le contexte d'une mutation de l'EGFR ou d'une translocation du gène ALK. La preuve de concept OSIRIS-LUNG vise également à démontrer la faisabilité de la mise en place d'une base de données standardisée contenant des données de vie réelle avec le modèle OSIRIS-FHIR entre deux centres de lutte contre le cancer et un hôpital universitaire.
Un modèle interopérable basé sur l’informatique décisionnelle
Conceptualiser le cancer pour mieux l’étudier sous un angle "data"
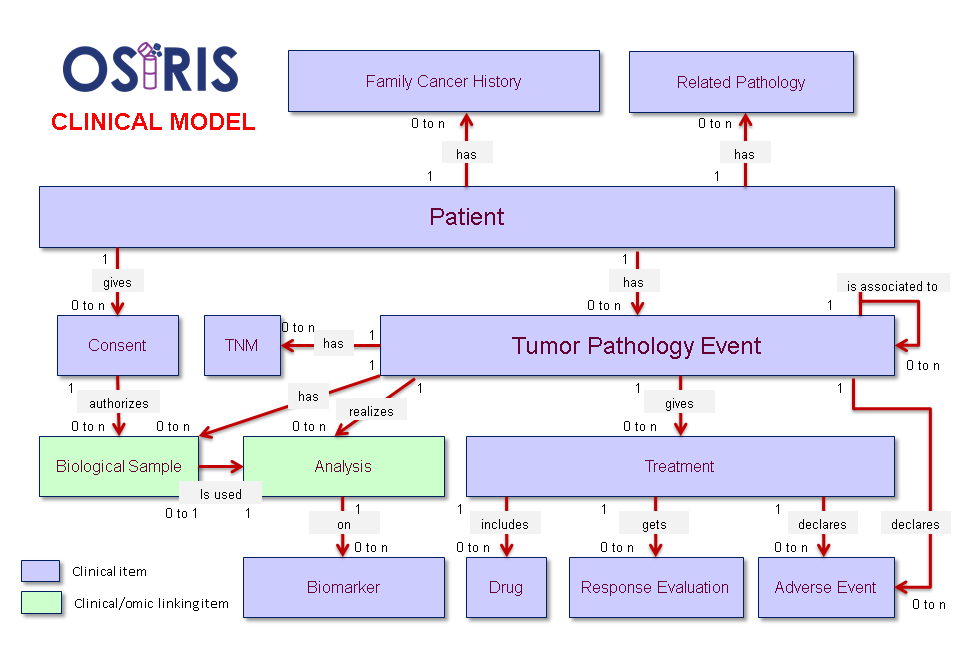
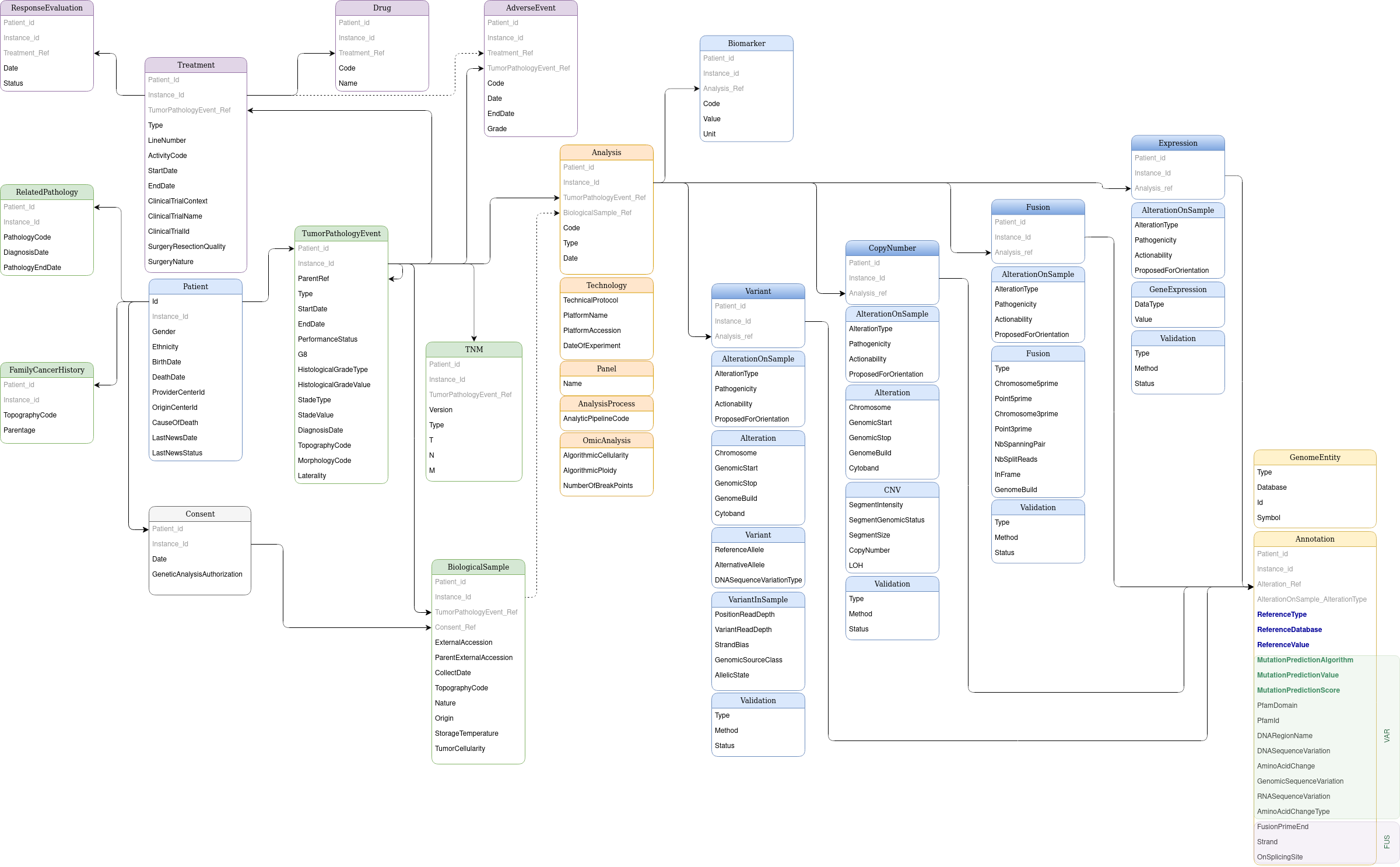
OSIRIS propose un modèle conceptuel de données conçu pour l’oncologie dans le cadre de l’exploitation de données cliniques et génomiques.
Le modèle clinique, centré sur le patient, prend en compte la dimension temporelle du cancer comme une somme d’évènements tumoraux. Ainsi chaque évènement tumoral implique des échantillons biologiques, des analyses (biomarqueurs, altérations génétiques, …) et un traitement.
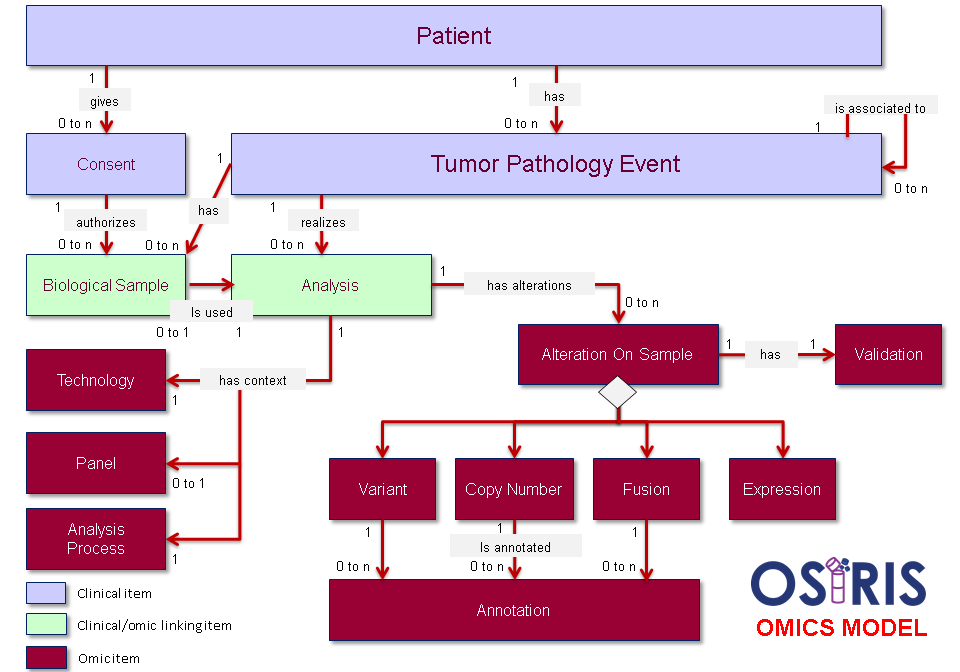
Le modèle génomique, centré sur l’analyse de l’échantillon biologique, caractérise d’une part la technologie analytique utilisée pour l’étude et d’autre part le type d’altération génétique (variant, nombre de copies, fusion, …).
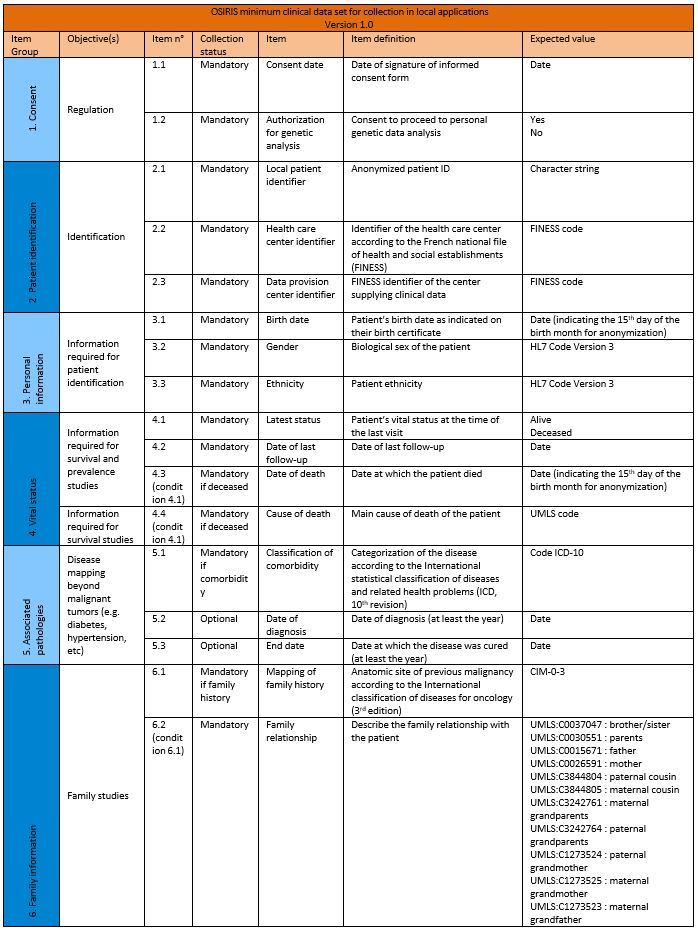
Un dictionnaire de données minimal pour l’oncologie : des terminologies et spécifications choisies pour la recherche
OSIRIS a été construit à partir de groupes de travail multidisciplinaires (cliniciens, data scientists, bio-informaticiens, radiologues, chercheurs) et la conceptualisation du cancer est ainsi exprimée à travers des variables médicales dans une terminologie internationale adaptée.
Un modèle logique et physique de données : une implémentation guidée au sein de votre système d’information hospitalier pour vos bases de données relationnelles
OSIRIS propose également un modèle logique et un modèle physique de données consistant à structurer les bases de données de recherche en oncologie au format OSIRIS dans le système de gestion de bases de données relationnelles.
Un modèle de recherche interopérable au format HL7 - FHIR : un échange de données possible au niveau national et international
FHIR est une norme pour l'échange de données sur les soins de santé, publiée par HL7®. Le modèle OSIRIS a ainsi été converti au format d’interopérabilité Health level 7 (HL7) FHIR (format JSON) afin de pouvoir échanger des données entre différents établissements de santé.
Un guide d’implémentation des ressources FHIR est disponible pour utiliser OSIRIS dans ce format d’interopérabilité et peut être consulté ici : https://fhir.arkhn.com/osiris/
Une gouvernance forte de l’Institut national du cancer
En 2023, l'Institut a repris le pilotage du projet OSIRIS.
Axes stratégiques
Ce travail est organisé selon quatre axes stratégiques :
- maintenir le modèle conceptuel de données OSIRIS, notamment en ce qui concerne les standards internationaux de santé (format interopérable OMOP (Observational medical outcomes partnership), ...) ;
- diffuser le modèle sur le territoire français, notamment en animant la « communauté OSIRIS » avec différentes interfaces web ou outils informatiques tels que GitHub ;
- étendre le modèle de données actuel d'OSIRIS, en particulier en ce qui concerne les données réelles extraites des dossiers médicaux électroniques et des logiciels cliniques ;
- mesurer l'impact de l'utilisation du modèle OSIRIS, à travers des problématiques scientifiques et des projets techniques.
Une utilisation fortement encouragée dans les appels à projets de l’Institut
L’Institut encourage l’utilisation du modèle conceptuel de données OSIRIS dans tous ses appels à projets nécessitant l’utilisation de données en oncologie.
De manière non exhaustive, la promotion du modèle concerne notamment les projets de recherche en pédiatrie.
Mettre en place OSIRIS au sein de votre système d’information hospitalier
De nombreux outils pour démarrer l’implémentation du modèle OSIRIS au sein de votre S.I.H. sont à votre disposition.
Une plateforme collaborative GitHub
L’Institut national du cancer administre un compte organisationnel sur la plateforme GitHub sur lequel l’ensemble des éléments du projet OSIRIS est à disposition de la communauté open source.
Chaque groupe multidisciplinaire travaillant sur une dimension du cancer peut ainsi déposer son code et son travail sur un FORK de la Main Branch qui constitue la version d’utilisation actuelle du package OSIRIS (Un dépôt secondaire de travail rattaché à un dépôt principal sur GitHub. Un FORK est un nouveau référentiel qui partage le code et les paramètres de visibilité avec le référentiel « en amont » d’origine).
Un guide d’implémentation HL7 FHIR
Pour les équipes qui souhaitent échanger des données de santé dans le cadre, un mapping vers les ressources FHIR actuellement proposé devra être réalisé.
Un guide d’implémentation permettra ainsi à l’utilisateur de mapper ses données issues du set OSIRIS vers le standard FHIR.
Un modèle physique de données
Afin d’utiliser le modèle théorique et conceptuel OSIRIS, un modèle physique de données est proposé pour structurer votre base de données de recherche en oncologie (clinique et génomique) au sein de votre système de gestion de base de données.
Des modèles de fichiers pivots
Le modèle théorique OSIRIS est en pratique décliné sous forme d’un modèle physique de données mais également sous forme de fichiers pivot au format CSV, fichiers qui peuvent être complétés par un processus d’Extraction, Transformation et Chargement (ETL) depuis votre entrepôt de données.
Des outils annexes disponibles pour la communauté
Un certain nombre d’outils complémentaires sont mis à disposition de la communauté et d’autres viendront s’ajouter au fur et à mesure de l’agrandissement de la communauté OSIRIS.
Des "requirements" au format txt sont ajoutés notamment pour guider l’utilisateur quant aux choix de versions de certains packages utilisés dans Python par exemple. Des jupyter notebooks sont également disponibles pour extraire par exemple l’ensemble des informations issues des fichiers d’imagerie au format DICOM (Digital Imaging And Communications in Medicine).
Pour toutes propositions d’améliorations ou retour d’expériences utilisateurs, merci de nous contacter par mail : osiris@institutcancer.fr ou d’ajouter une issue sur la FORK concernée.
En savoir plus : la fiche d'identité d'OSIRIS sur le Health Data Hub